원하는 target gene의 발현을 확인하기 위해서는 유전자에 맞는 primer가 필요합니다.
숙달되신분들은 쉽게 만들겠지만 저는 초보자를 위한 방법을 적겠습니다.
순서대로 따라오시면 누구나 하실수 있습니다. 화이팅!
우선, 분석하려는 유전자의 variant들이 몇개씩 있습니다. 공통된 부분의 sequence를 찾아야지, 특정 variant의 sequence를 타겟하면 그것만 선택적으로 detection 하게 됩니다.
순서는 다음과 갔습니다.
1. target gene의 variant들의 공통적인 sequence를 찾아야합니다.
2. 공통적인 sequence를 이용하여 primer를 제작합니다.
1번이 조금 시간이 걸리지만 blast를 이용해서 쉽게 찾을 수 있고. 2번 같은 경우는 업체에서 제공하는 프로그램으로 sequence만 넣으면 끝납니다.
1. Nucleotide를 선택하시고 target gene을 검색합니다.

2. 왼쪽의 mRNA를 check해주시고, 오른쪽에 Organisms을 선택합니다. 보통은 휴먼 세포를 사용하므로 Homo sapiens를 선택했습니다.

3. 각 gene마다 variant가 다른데, 모두 봐줘야 합니다.
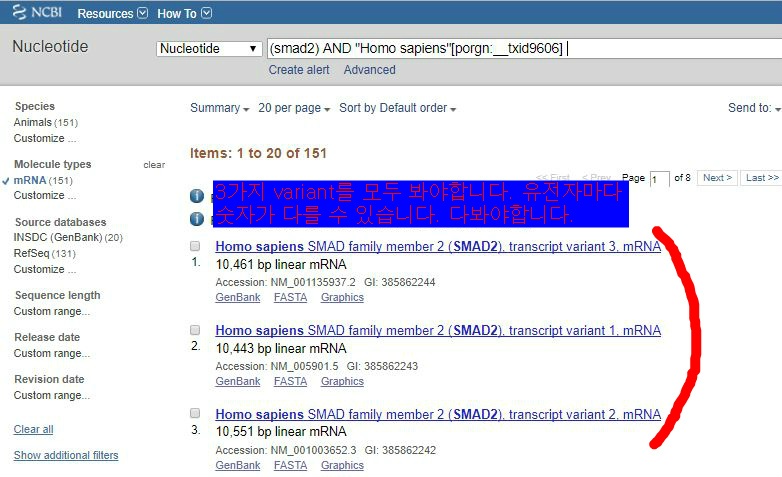
4. 먼저 variant 1을 클릭하여 들어갑니다. 그러면 유전자에 관한 정보가 많이 나옵니다. 이중에서 우리 찾아야 할 것은 CDS입니다.

5. 밑으로 내리면 CDC를 찾으실 수 있습니다. 이것은 유전자의 긴 sequence중에서 protein으로 translation되는 부분을 나타냅니다.

6. CDS를 클릭하면 밑으로 자동으로 내려갑니다. 그리고 아래 그림처럼 갈색으로 protein으로 translation되는 sequence가 표시 됩니다. 저 부분을 드래그해서 복사합니다.

7. variant 1에서 3까지 동일한 과정을 반복합니다. 그리고 복사한 것은 보기 쉽게 word에 복사해서 아래처럼 붙입니다.

8. 다음 할 것은 3개의 sequence를 비교하여 공통된 부분을 찾는 것입니다. 눈으로보면 절대 같은 곳을 못찾죠. 그래서 NCBI 사이트에 맨 아래를 보면 BLAST가 있습니다. BLAST는 sequence를 비교 분석해주는 툴입니다.
지금까지 쉽죠?^^
9. BLAST에 들어오면 여러가지 있는데 그중에 Nucleotide BLAST를 누르시고 들어가면 됩니다.
10. 그리고 2가지 sequence를 비교하기 위해서는 아래의 그림에서 보는 거와 같이 Align two or more sequences를 클릭해줍니다.
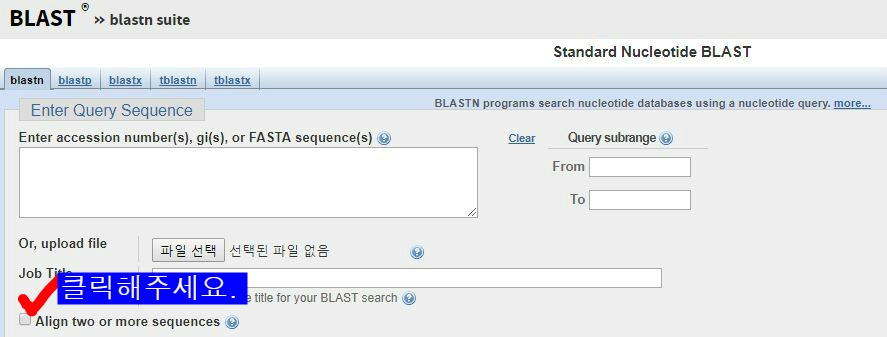
11. 그러면 sequences를 넣을 수 있는 칸이 하나 더 생깁니다.
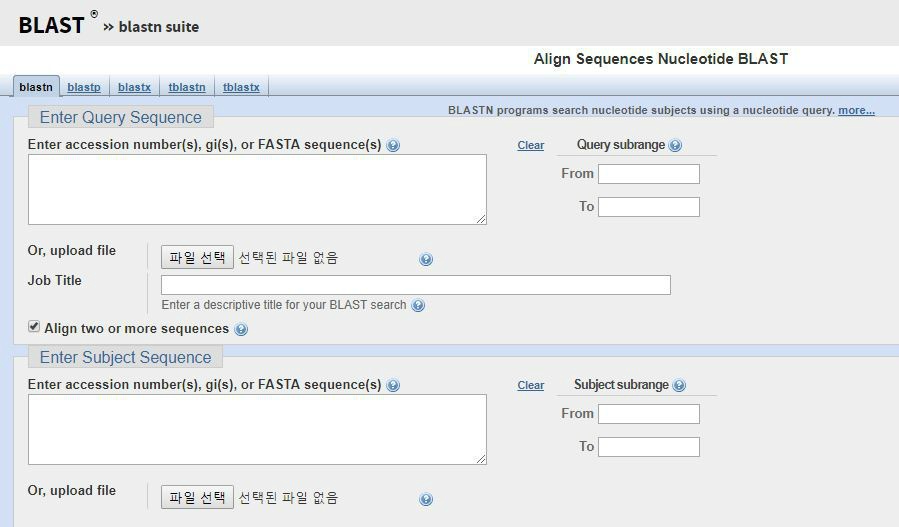
12. 첫 칸은 variant 1 sequence를 두번째 칸은 variant 2 sequence를 넣어 줍니다.
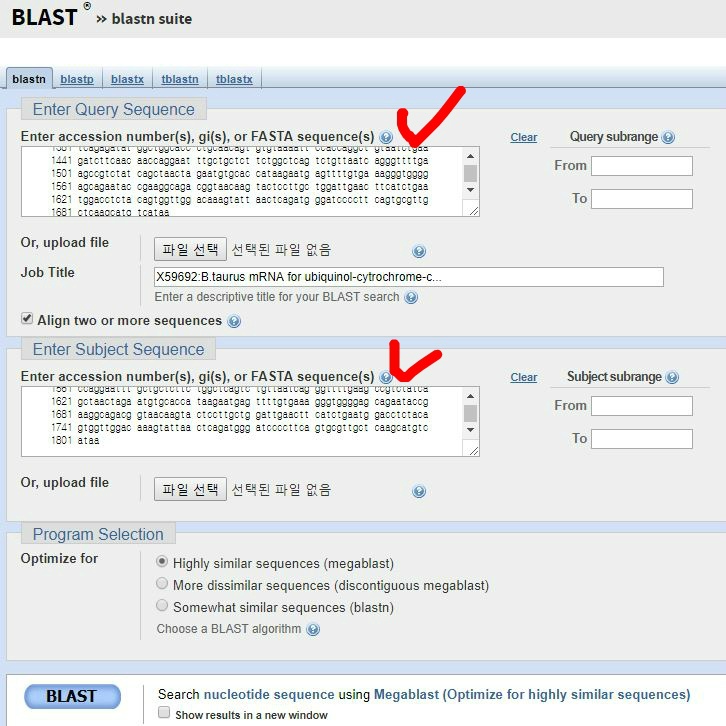
13. BLAST를 눌러주면 아래와 같이 매치되는 sequence를 보여줍니다. 굉장히 복잡하죠? 아래그림은 variant 1과 2가 100% 일치한다는 결과 입니다.
혹시 다르면 word 프로그램에서 ctrl+F 로 맨 앞 ATGTC를 찾아 표시하고 맨 뒤의 GAAA를 찾아서 표시해주면서 공통된 부분을 찾아줍니다.

14. variant 1과 3은 차이가 있는데요. 그러면 갈색 이외의 색으로 표시하면 됩니다.
variant 1 vs variant 2 -> variant 2 vs variant 3 -> variant 3 vs variant 1
모두 비교해주면 공통된 sequence를 찾을 수 있습니다.

이제 다왔습니다. 다음 단계는 간단합니다. 다른 방법도 있겠지만 제가 사용하는 방법을 소개하겠습니다.
구글에서 <Oligoperfect Designer>를 검색해줍니다. Thermo Fisher를 광고하는 것은 절대 아니예요.
**220225 업데이트**
써모피셔에 이제는 가입해야 oligoperfect를 사용할 수 있습니다. 가입해서 사용하시면 아래이어서 하는 설정없이 프라이머를 제작할 수 있습니다. 그리고 써모에서 제공하는 SEQUENCE는 specificity를 꼭 확인해서 주문하시길 바랍니다.
1. Sequence name과 Researcher Name은 아무거나 써도 상관 없습니다. Application은 PCR: Detection을 선택해줍니다.
Target Sequence에서는 앞에서 분석한 공통 부분의 sequence를 복사해서 넣습니다.
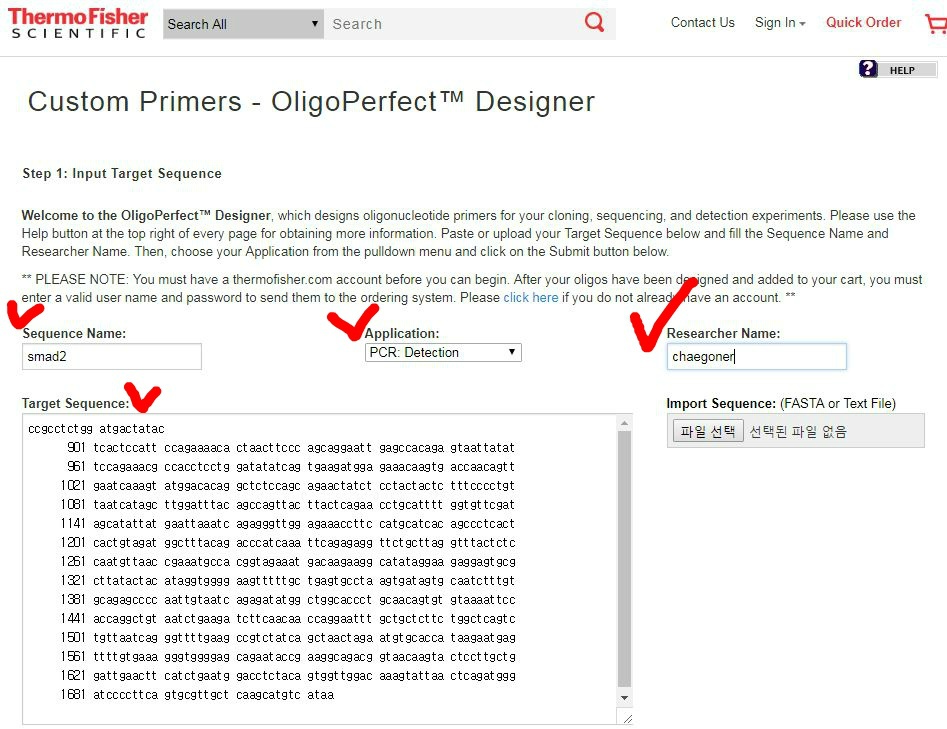
2. Submit을 눌러주시면 조건들이 나옵니다. 아래에 check한 부분만 그림처럼 바꿔주면 됩니다. 그리고 다시 Submit을 누릅니다.
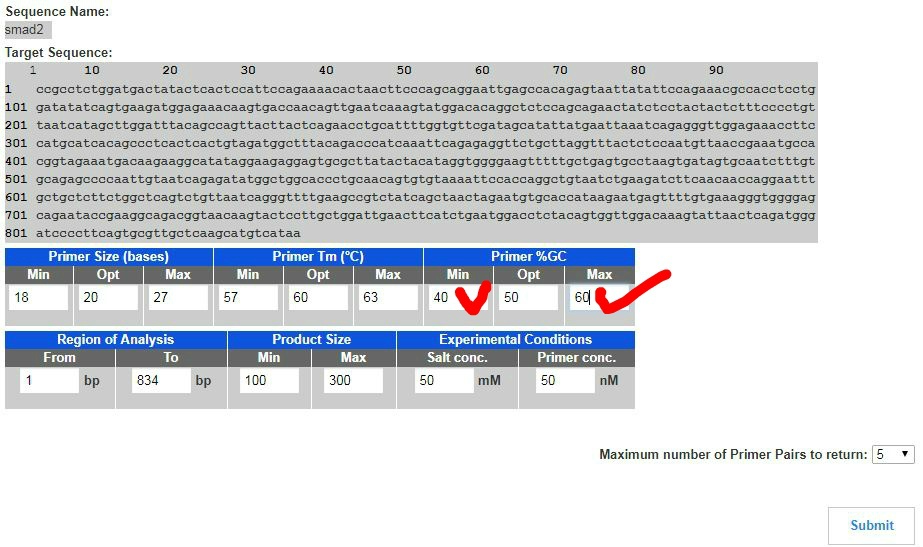
3. 5개 정도의 후보가 나오는 것을 볼 수 있습니다. 그중에 c-terminal 쪽에 가까운 primer를 선택하시면 됩니다 .
아래에 보시면 Primer sequence 두 개가 나오는걸 보실수 있습니다. forward랑 backward 둘다 복사해서 코스모진텍 같은 primer 만들어주는 회사테 합성해달라고 하면 됩니다.
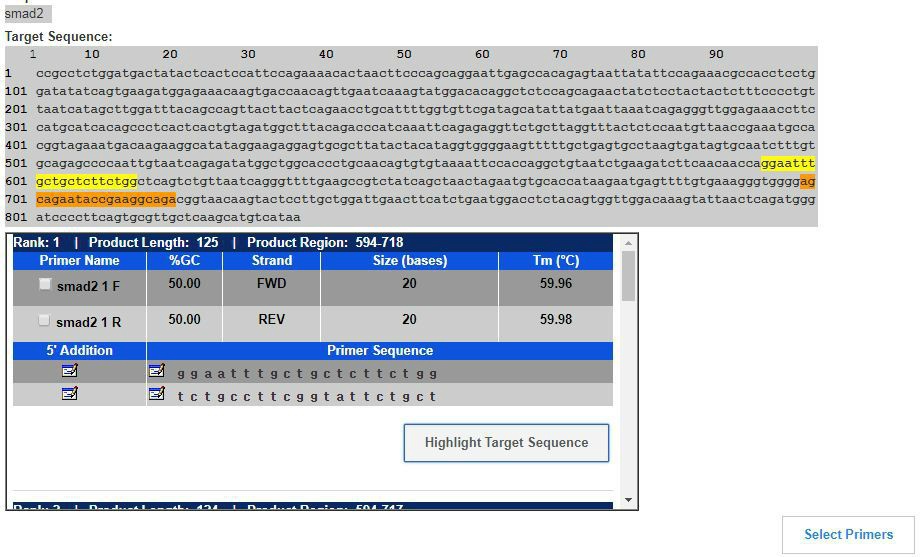
끝!! 쉽죠?!
처음 해보면 잘 모를수도 있겠지만 차근차근 해보세요. 그리고 혹시 이해 안되는 부분이 있다면 댓글로 알려주세요. 가르쳐드리겠습니다.
![]()
많은 분들이 제작한 primer의 specificity를 확인하는 방법을 문의하셔서 이후 과정을 다시 포스트 했습니다.
아래과정까지 꼭 확인 해보시기 바랍니다.

<댓글과 공감은 작성자에게 큰 도움이 됩니다.>
'난분자생물학전공자_아무것도모르지' 카테고리의 다른 글
| 데자뷰란? 데자뷰의 과학적인 원인과 이유 (0) | 2023.06.03 |
|---|---|
| 스피어만 상관분석 (Spearman's correlation) 방법 (2) | 2022.01.10 |
| 인류의 운명은 어떻게 결정되었는가 - 총,균,쇠 (0) | 2022.01.10 |
| Immune cell surface 마커 종합 (0) | 2021.05.28 |